Proteômica GenOne
Além dos serviços de Genômica avançados oferecidos pela GenOne, também oferecemos serviços de análise de proteínas. Juntas, Genômica, Transcriptômica e Proteômica, permitem um alto nível de compreensão dos sistemas biológicos.
 Perfil Proteômico
Perfil Proteômico
O perfil proteômico é estudo com o objectivo de caracterizar o número máximo possível de proteínas de um organismo completo, fluidos corporais, ou extratos.
A criação deste perfil pode servir de base para futuros estudos de alterações quantitativas ou avaliações de proteínas específicas. Em combinação com a análise transcriptômica, os perfis proteômicos fornecem informações claras sobre a dinâmica de um dado sistema biológico.
Casos:
The Oyster Genome Reveals Stress Adaptation and Complexity of Shell Formation. Nature. 490, 49–54 (2012).
In-depth Proteome Analysis of The Rubber Particle of Hevea Brasiliensis (Para Rubber Tree). Plant Mol Biol. 82(1-2):155-68 (2013).
Proteômica Quantitativa – iTRAQ.
 A tecnologia iTRAQ (isobaric tag for relative and absolute quantitation), é uma técnica não baseada em eletroforese em gel que é utilizada para quantificar proteínas de diferentes fontes de uma única vez. O método baseia-se na marcação covalente da porção N-terminal e das aminas de cadeia lateral de peptideos a partir da digestões de proteínas com tags de massa variável.
A tecnologia iTRAQ (isobaric tag for relative and absolute quantitation), é uma técnica não baseada em eletroforese em gel que é utilizada para quantificar proteínas de diferentes fontes de uma única vez. O método baseia-se na marcação covalente da porção N-terminal e das aminas de cadeia lateral de peptideos a partir da digestões de proteínas com tags de massa variável.
O iTRAQ_8-plex_kit que nós fornecemos podem ser usados para marcar todos os péptidos de diferentes amostras/tratamentos. Estas amostras são então reunidas e geralmente fraccionadas por nano cromatografia líquida, seguido de análise por espectrometria de massa em tandem (MS/MS). Uma busca em bancos de dados é então realizada utilizando os dados da fragmentação para identificar os peptidos marcados e, consequentemente, as proteínas correspondentes. A fragmentação do tag gera uma baixa massa molecular de íons repórters que podem ser utilizada para quantificar relativamente os peptideos e as proteínas que lhes deram origem.
Casos:
Identification of Novel Biomarkers for Sepsis Prognosis via Urinary Proteomic Analysis Using iTRAQ Labeling and 2D-LC-MS/MS. Plos one. DOI: 10.1371/journal.pone.0054237 (2013).
Proteômica Quantitativa – Label-Free
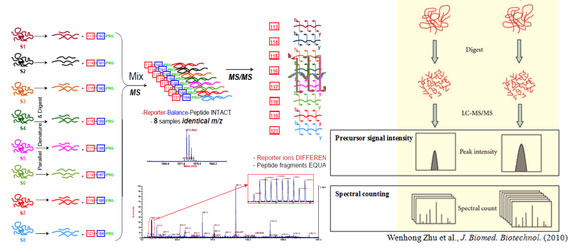
 Esta técnica é baseada na intensidade do sinal precursor ou na contagem espectral. O primeiro é na maioria dos casos aplicados a dados adquiridos em espectrômetros de massa de alta precisão, equipados com analisadores de massa de nova geração. A energia de alta-resolução facilita a extração de sinais dos peptídeos no nível MS1 e assim desacopla a quantificação do processo de identificação. Com a contagem espectral ocorre a quantificação do número de espectros identificados para um dado peptídeo em diferentes amostras biológicas e, em seguida, integra os resultados para todos os peptídeos obtidos da(s) proteína(s) que são quantificadas.
Esta técnica é baseada na intensidade do sinal precursor ou na contagem espectral. O primeiro é na maioria dos casos aplicados a dados adquiridos em espectrômetros de massa de alta precisão, equipados com analisadores de massa de nova geração. A energia de alta-resolução facilita a extração de sinais dos peptídeos no nível MS1 e assim desacopla a quantificação do processo de identificação. Com a contagem espectral ocorre a quantificação do número de espectros identificados para um dado peptídeo em diferentes amostras biológicas e, em seguida, integra os resultados para todos os peptídeos obtidos da(s) proteína(s) que são quantificadas.
Casos:
Comparative Analysis of Dynamic Proteomic Profiles between in Vivo and in Vitro Produced Mouse Embryos during Postimplantation Period. J. Proteome Res. 12 (9), 3843–3856 (2013).
Identificação de Proteínas
 A identificação de proteínas pode ser executada em uma grande variedade de amostras, incluindo spots ou bandas de gel, IP, etc. As proteínas são analisadas tanto por MALDI-TOF/TOF ou por nano-LC-ESI-MS/MS e, em seguida, identificados utilizando as massas peptídicas ou espectros de fragmentação MS/MS.
A identificação de proteínas pode ser executada em uma grande variedade de amostras, incluindo spots ou bandas de gel, IP, etc. As proteínas são analisadas tanto por MALDI-TOF/TOF ou por nano-LC-ESI-MS/MS e, em seguida, identificados utilizando as massas peptídicas ou espectros de fragmentação MS/MS.
Nós fornecemos os resultados de identificação de proteínas em um relatório eletrônico que também inclui os resultados da análise bioinformática, como GO e vias de sinalização.
Serviços:
Identificação de proteínas em spots/bandas de géis
Identificação de proteínas de bandas visíveis ou spots obtidas de géis corados com Coomassie ou ou com prata.
- Identificação de proteínas desconhecidas em géis 1D ou 2D.
- Verificação de produtos de expressão de proteínas.
- A detecção de proteínas co-preciptadas em testes de imunoprecipitação.
Casos:
Proteomics Study of Rice Embryogenesis: Discovery of The Embryogenesis-dependent Globulins. Electrophoresis. 33(7):1129-38 (2012).